https://pubmed.ncbi.nlm.nih.gov/33638807/
Los coronavirus son virus de ARN que pueden causar variedad de complicaciones en la salud humana e incluso la muerte. A la fecha, se han identificado 3 coronavirus capaces de infectar humanos: coronavirus del síndrome respira-torio agudo severo (SARS-Co-V), SARS-CoV-2 y el coronavirus del síndrome respiratorio de Oriente Medio (MERS-CoV).
El SARS-CoV-2 ha infectado aproximadamente a 100 millones de personas y causado 2 millones de muertes en todo el mundo. Mientras que algunos pacientes con COVID-19 permanecen asintomáticos o presentan síntomas leves similares a los de la gripe, otros desarrollan dificultad respiratoria grave, complicaciones cardíacas, insuficiencia renal, cho-que séptico y otras complicaciones de salud a largo plazo.
En este contexto de pandemia por COVID-19, se ha trabajado en la búsqueda de nuevas opciones terapéuticas de tratamiento antiviral. Las estrategias terapéuticas actuales para tratar a los pacientes con COVID-19 son sintomáticas, y en casos graves se administra el fármaco antiviral remdesivir.
Entre los esfuerzos actuales para combatir el COVID-19, algunos enfoques incluyen la búsqueda de inhibidores de entrada y replicación y la reutilización de medicamentos antivirales existentes. Hasta la fecha, se han registrado más de 2000 ensayos clínicos de COVID-19 en todo el mundo, que van desde enfoques de moléculas pequeñas (incluidos fármacos como lopinavir-ritonavir) hasta terapias con plasma de convalecencia y células madre
Además, diferentes organismos reguladores de todo el mundo han autorizado el uso de varias vacunas COVID-19. Sin embargo, a pesar del desarrollo de vacunas efectivas, las investigaciones continuas sobre todos los enfoques terapéuticos siguen siendo cruciales, especialmente en el contexto de las variantes emergentes del SARS-CoV-2 y las posibles cepas resistentes a las vacunas.
Una estrategia antiviral relevante para el SARS-CoV-2 podría provenir de la respuesta antiviral natural del huésped. Las células hospedadoras pueden desarrollar diferentes estrategias antivirales contra los virus de ARN, y una estrategia eficaz es la interferencia de ARN.
La interferencia de ARN incluye un grupo de moléculas conocidos como microARN (miARN / miR-), que son moléculas de ARN no codificantes, típicamente de 20-25 nucleótidos de longitud, que regulan la expresión génica, la señalización celular y el entorno de la célula huésped.
Actuando como reguladores de genes postranscripcionales, los miARN se emparejan con el 3′-UTR de secuencias de ARNm complementarias para mediar en su degradación o inhibir su traducción. También se ha informado que las interacciones miRNA-mRNA no canónicas, como las que interactúan con el 5′-UTR o las regiones codificantes de los genes diana, son funcionales.
Dado que se han identificado aproximadamente 2000 miRNAs humanos (miRBase 22), y se ha predicho que regulan más del 60% de todos los genes codificadores de proteínas humanas, los miRNAs sirven como una vía plausible para la investigación terapéutica, especialmente para la modulación de proteínas que no pueden ser dirigidas por otras moléculas pequeñas.
Hay dos enfoques generales para desarrollar terapias basadas en miARN (Figura 1). El primero se basa en el desarrollo de antagonistas o inhibidores de miARN para aumentar o rescatar la expresión de proteínas específicas que están reguladas negativamente. El segundo enfoque consiste en el uso de imitadores de miARN para regular negativamente la expresión de proteínas inducidas anormalmente. En los últimos años, varios tratamientos basados en miARN se han mostrado prometedores en ensayos clínicos.
Por ejemplo, se han desarrollado fármacos para la infección por el virus de la hepatitis C (VHC) que actúan como antagonistas de miRN-22, reduciendo los niveles de ARN viral al secuestrar el miRN-122 del genoma viral, donde el virus lo utiliza para mejorar su propagación.

Figura 1. Posibles aplicaciones terapéuticas de los miARN.
También se ha informado de que los miARN tienen un papel en el interrogatorio de las interacciones entre el huésped y el virus durante la infección viral. Ya sea a través de interacciones directas con el genoma viral o induciendo respuestas celulares antivirales en el huésped, los miARN sirven como reguladores críticos de la patogénesis viral (Figura 2).
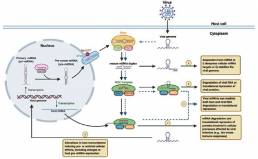
Figura 2. Posibles interacciones entre los miARN y la infección viral.
También se ha demostrado que los virus crean microambientes específicos que facilitan el ciclo de vida viral a través de alteraciones en la expresión de miARN del huésped. Esta comunicación cruzada entre los miARN del hospedador y los virus puede alterar el transcriptoma del hospedador y regular indirectamente las infecciones virales mediante la modulación de las vías celulares del hospedador, lo que da como resultado efectos pro o antivirales generales.
Por ejemplo, un estudio encontró que la expresión elevada de miR-155 en linfocitos infectados con el virus de Epstein-Barr (EBV) contribuyó a un estado celular proviral general al regular negativamente simultáneamente el factor nuclear kappa-potenciador de la cadena ligera de las células B activadas (NF-κB) la señalización, que es importante para la producción de citocinas y la supervivencia celular, y la supresión de las respuestas inmunitarias innatas a la infección viral].
De hecho, se ha demostrado que los miARN regulan la respuesta inmune innata del huésped a las infecciones virales mediante una variedad de mecanismos, que incluyen la alteración del microambiente lipídico, la promoción de vías de señalización celular que conducen a la expresión de genes antivirales inducibles por interferón (INF), o mejorando la expresión de los reguladores de la respuesta inmunitaria
Un estudio identificó miR-24, miR-124 y miR-744 como miARN antivirales de amplio espectro contra el virus de la influenza A (VIA) y el virus respiratorio sincitial (VRS). Al dirigirse a la vía de señalización de la proteína quinasa activada por mitógenos p38 (MAPK), estos miARN regulan la producción de citocinas involucradas en la respuesta inmune, así como la entrada y propagación viral, contribuyendo a un fenotipo antiviral general.
Se ha demostrado que los miARN antivirales inducidos por el VHC se dirigen al ARN viral e inhiben su replicación, mientras que otros miARN, como el miR-135a, mejoran la replicación viral a través del antagonismo de las vías antivirales celulares.
Evidentemente, como lo establecen muchos informes, las interacciones miARN-virus son extensas y complejas y poseen la capacidad de mejorar o suprimir la replicación viral.
Con respecto a la infección por VHC, se ha demostrado que los miARN antivirales inducidos por IFN se dirigen al ARN viral e inhiben su replicación, mientras que otros miARN, como el miR-135a, mejoran la replicación viral a través del antagonismo de las vías antivirales celulares.
Los miARN del huésped también pueden interactuar directamente con el genoma viral para regular las infecciones virales. Con el fin de mejorar la replicación viral, algunos virus utilizan miARN derivados del hospedador para estabilizar su genoma y prevenir la degradación.
Un ejemplo de esto incluye el proviral miR-122 mencionado anteriormente, que se une directamente al 5′-UTR del VHC y estabiliza eficazmente el ARN para promover la replicación viral. Curiosamente, el genoma del VHC también puede actuar como una «esponja» para secuestrar miR-122 lejos de sus objetivos celulares, proporcionando así otro mecanismo para mejorar su potencial infeccioso.
Otros virus, como algunos herpesvirus, también utilizan este método de secuestro de miARN para des-reprimir dianas celulares como estrategia viral para modular la célula huésped.
Por el contrario, también se ha sugerido que los miARN celulares pueden interactuar con el genoma viral para inhibir la replicación. Por ejemplo, en la infección por el virus de la inmunodeficiencia humana 1 (VIH-1), se descubrió que un grupo de miARN celulares induce la latencia viral a través de interacciones con los extremos 3 ‘del ARN viral. Además de los miARN derivados del hospedador, también se han identificado miARN codificados por virus (o miARN virales) en varios virus, incluido el VEB humano.
Al explotar la maquinaria de miARN celular del huésped, los miARN virales pueden expresarse para modular tanto los objetivos de ARN del huésped como del virus, y algunos incluso ayudan al virus a evadir la respuesta inmune del huésped. En general, al hacer que la célula huésped sea más susceptible a la infección viral o mediante interacciones directas con el virus, los miARN desempeñan un papel importante en la regulación de las infecciones virales y se pueden explorar para desarrollar nuevos antivirales.
Evidentemente, los miARN pueden regular una amplia variedad de infecciones virales, incluidas infecciones respiratorias como el VHC. Por ejemplo, un estudio realizado por Mallick et al. encontraron que las proteínas virales N y S del SARS-CoV regulaban negativamente miR-223 y miR-98, respectivamente, con el fin de modular la diferenciación de la célula huésped e inducir respuestas proinflamatorias como el aumento de la activación de quimiocinas inflamatorias. Si bien se han establecido interacciones entre los miARN y el SARS-CoV, aún no se comprende completamente la capacidad de los miARN para regular el SARSCoV-2. Dicho esto, varios grupos han realizado recientemente investigaciones computacionales sobre objetivos de miARN en el genoma del SARS-CoV-2; sin embargo, aún se requiere una mayor validación experimental para validar estas predicciones y determinar sus efectos celulares posteriores. Esta laguna en el conocimiento, junto con las opciones de tratamiento limitadas para el SARS-CoV-2, resalta la importancia de una mayor investigación sobre las interacciones de miARN y SARS-CoV-2.
En este estudio se utilizó la herramienta de búsqueda de alineación local básica de nucleótidos (BLASTn) para secuencias muy similares, para identificar posibles sitios de unión de miARN en los genomas virales de SARS-CoV-2, SARSCoV y MERS-CoV. Se identificaron miARN con un 100% de complementariedad del sitio de siembra con el genoma viral y luego se realizaron más investigaciones bibliográficas sobre sus posibles funciones en la infección por SARS-CoV-2.
Si bien los estudios sobre las interacciones entre los miARN del huésped y los genomas virales son vitales, simplemente reflejan un paso inicial en el desarrollo de terapias antivirales basadas en miARN.
Identificación de microARN con posibles funciones en la infección por SARS-CoV-2.
Considerando el potencial establecido para que los miARN influyan en las infecciones virales (ya sea con efectos pro o antivirales), se investigaron las interacciones entre los miARN humanos y el genoma del SARS CoV-2 para obtener información y dirigir el desarrollo de miARN basado en terapéutica (Figura 3A). En este enfoque, se optó por observar los miARN más comúnmente expresados en tejidos y tipos de células a menudo atacados por el virus, como una forma de identificar miARN que pueden modificar el microambiente celular del huésped para que sea más favorable para la infección viral.
Se utilizó la base de datos de nucleótidos NCBI para obtener las secuencias del genoma viral para SARS-CoV-2 (MN908947.3), SARS CoV (NC_004718.3) y MERS-CoV (NC_019843.3). Usando la base de datos mimiRNA, el centro de este estudio estuvo en los 20-30 miRNAs expresados de forma más significativa en células epiteliales pulmonares A549, corazón, hepatoma (hígado) e intestino delgado como punto de partida. A partir de los resultados obtenidos, se seleccionaron los 12 miARN principales que estaban presentes en todos los tipos de células y muy abundantes en las células A549 del epitelio pulmonar para su posterior investigación.
Se identificaron miARN adicionales a través de una revisión de la literatura de miARN que se demostró que tienen un papel en la regulación del metabolismo, la respuesta inmune y otras infecciones virales.
Las secuencias de la hebra guía para los miARN humanos (hsa) identificados para la investigación se obtuvieron a partir de miRBase. Para cada miARN, se determinó la región semilla (nucleótidos 2-8) y su correspondiente secuencia complementaria. Se utilizó BLASTn para secuencias muy similares para buscar coincidencias entre las secuencias del genoma viral (SARS-CoV-2, SARS-CoV y MERS-CoV) y las secuencias de semillas de miARN complementarias. Se predijeron los posibles sitios de unión de miARN en ubicaciones con 100% de complementariedad. Los sitios de unión predichos en el genoma del SARS-CoV se cotejaron con la base de datos ViTa utilizando la cepa TWH del SARSCoV. Para cada sitio de unión potencial, se identificó la ubicación y la región correspondiente en el genoma viral.
También se utilizó miRDB, DIANA microT-CDS y el software de predicción TargetScan para predecir los miRNA que se dirigen al 3′-UTR del receptor ACE2 en humanos (NM_021804.3) para explorar si los miRNA podrían ser buenos objetivos para el tratamiento de la infección por SARS-CoV-2.
Las búsquedas incluyeron sitios altamente y mal conservados, así como sitios de unión 6mer, 7mer, 8mer y 9mer. Los principales éxitos generados aquí proporcionan un punto de partida ideal para más análisis de expresión de miARN-ACE2 que, a su vez, pueden ayudar a dirigir el desarrollo de terapias antivirales con miARN.
(Figura 3b).
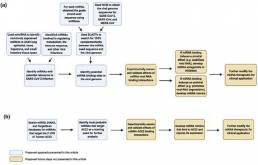
Figura 3. Descripción general del enfoque terapéutico antivírico basado en miARN propuesto contra la infección por SARS-CoV-2.
Posibles funciones y sitios de unión de microARN en los genomas del SARS-CoV-2, SARS-CoV y MERS-CoV
Entre los 39 miARN investigados, se predijo que 29 tenían sitios de unión dentro del genoma del SARS-CoV-2 en función de la complementariedad del sitio de semillas.
Se predijo un gran número de estos sitios de unión en el ORF1ab, ya que es la región más grande del genoma, abarca aproximadamente 21 kb de longitud y codifica numerosas proteínas virales. No se predijeron sitios de unión de miARN en las UTR de SARS-CoV-2 y SARS-CoV. Esto sugiere que los miARN investigados en este estudio pueden no unirse directamente al genoma viral para estabilizar o promover la degradación del ARN viral; sin embargo, los sitios de unión en las regiones codificantes pueden seguir siendo funcionales. Dado que solo se utilizó una secuencia genómica para evaluar cada virus, no se capturaron las variaciones en los genomas virales que pueden permitir sitios de unión de miARN adicionales. Además, el hecho de que los sitios de unión de miARN en los genomas virales se definieran como ubicaciones con un 100% de complementariedad del sitio de siembra es bastante restrictivo, ya que se sabe que los miARN pueden unirse a sitios de manera imperfecta.
Esto lleva a creer que es posible que no se hayan representado otros sitios de unión potenciales. Además, estos miARN celulares, con o sin sitios de unión en el genoma viral, aún pueden tener un papel en la regulación de vías inflamatorias o de señalización en el huésped, como se observa con otras infecciones virales.
En términos del genoma de MERS-CoV, se predijo que miR-92 se dirigiría a la 3′-UTR, mientras que se predijo que miR-130 y miR-199 se dirigirían a la 5′-UTR. Si bien la regulación de MERS-CoV mediada por miARN del huésped no está bien explorada, es interesante señalar que miR-199a se ha identificado previamente para regular la expresión de TMPRSS2 en el hígado, el estómago y el cuerpo uterino, ya que esta proteasa es fundamental para la entrada del SARS-CoV2, SARSCoV y MERS-CoV en las células. Por lo tanto, tras un análisis adicional y una validación experimental para determinar si estos son sitios de unión de miARN verdaderos, se pueden explorar las funciones de estos miARN en la modulación de la infección por MERS-CoV, junto con otros virus relacionados.
El número relativo y las ubicaciones de los posibles sitios de unión de miARN en el genoma también pueden proporcionar una idea de las funciones que estos miARN pueden desempeñar durante las infecciones virales.
A partir de este análisis, se encontró que miR-16 tiene el mayor número total de sitios de unión en los tres genomas virales, con 21 sitios de unión en el genoma de SAR-CoV, 15 sitios de unión en el genoma de SARS-CoV-2 y 14 de unión sitios en el genoma de MERSCoV (Figura 4). El hecho de que el elevado número de sitios de unión de miR-16 sea común entre los tres VHC podría indicar que estos virus han desarrollado mecanismos provirales que utilizan este miRNA para mejorar su propagación, como se observó previamente en el VHC. De manera similar, también se encontró que miR-29 y miR-30 tienen un gran número de sitios de unión potenciales, con diez sitios de unión predichos cada uno en el genoma del SARS-CoV-2. En términos de infección por SARS-CoV-2, se identificaron además que miR-200 y miR-24 son de particular interés, ya que se ha demostrado que están vinculados a la regulación de ACE2 y furina, respectivamente (Figura 5).
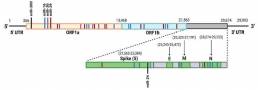
Figura 5. Representación esquemática de los sitios de unión de miARN predichos en el genoma del SARS-CoV-2.
5 microARN que regulan la expresión del receptor ACE2
Además de los miARN que interactúan directamente con el genoma viral, los miARN del huésped que se dirigen a ACE2 también pueden participar en la regulación de la infección por SARS-CoV-2. Como ACE2 es el receptor viral que facilita la entrada del SARS-CoV-2 en las células, la comprensión de qué miARN modulan su expresión puede proporcionar información para posibles tratamientos antivirales basados en miARN. En este estudio, se identificaron numerosos miARN que se dirigen a la 3′-UTR de ACE2 en humanos. Se predijó un total de seis miRNA (miR-362-5p, miR-421, miR-500a-5p, miR-500b-5p, miR-3909 y miR-4766-5p) mediante las tres herramientas de predicción de miRNA en línea. Curiosamente, miR-421 también se ha identificado previamente como un posible regulador de ACE2, lo que lo convierte en un objetivo plausible para una mayor investigación terapéutica contra el SARS-CoV-2.
Abundantes microARN cuya función normal puede ser modulada por el SARS-CoV-2
En la mayoría de los casos, los miARN regulan la expresión del gen diana uniéndose a la 3′-UTR de los ARNm; sin embargo, los genomas virales son significativamente más grandes que el ARNm normal, por lo que su regulación e interacciones con los miARN pueden ser más complicadas. Se ha demostrado que los sitios de unión de miARN en las UTR 5 ‘y 3’ y las regiones codificantes pueden ser funcionales. Si bien no se predijeron sitios de unión en el 3′-UTR del genoma del SARS-CoV-2 en este análisis, se identificaron tres miARN con sitios de unión predichos en las regiones codificantes del SARS-CoV-2 (miR-16, miR-200 y miR-24).
Funciones y vías de miR-16 que pueden verse afectadas durante la infección por SARS-CoV-2
El alto número de sitios de unión de miR-16 predichos en los tres genomas del VHC sugiere que el ARN viral puede actuar como una «esponja» para secuestrar miR-16, disminuyendo así su abundancia en las células infectadas y provocando una desrepresión de sus objetivos en el huésped. Como se determinó a partir de estudios anteriores, una función de miR-16 es inducir la apoptosis regulando a la baja el factor de supervivencia BCL2. Se ha indicado que la apoptosis es una parte importante de la respuesta inmune antiviral innata en algunas infecciones virales. Por ejemplo, en células de ratón infectadas con influenza, se encontró que la apoptosis era inducida y posteriormente las células apoptóticas eran el objetivo de los macrófagos y neutrófilos para la fagocitosis. Al regular a la baja miR-16, los virus como el SARS-CoV-2 pueden evadir este mecanismo inmune innato al suprimir la apoptosis de la célula huésped.
También se ha demostrado que la supresión de miR-16 aumenta la producción de especies de oxígeno reactivas mitocondriales (ROS) y la expresión del receptor tipo Toll 4 (TLR4).
Esto es de particular interés ya que tanto la producción de ROS como la expresión de TLR4 pueden tener efectos negativos en las células pulmonares e incluso causar daño pulmonar, algo que es pertinente en virus respiratorios como el SARS-CoV-2 y el SARS-CoV. La producción de ROS desencadena una reacción a través de la vía de señalización TLR4-TRIF que, en última instancia, aumenta la producción de citocinas en los macrófagos pulmonares, lo que provoca inflamación y daño en los pulmones. También se ha demostrado que el desequilibrio redox resultante de la infección por SARS-CoV y el aumento de la producción de ROS inducen la expresión del factor de crecimiento transformante beta 1 (TGF-β1), una citocina profibrogénica que aumenta aún más la producción de ROS, y se ha relacionado con el desarrollo de lesiones y tejido pulmonar cicatrizado, comúnmente denominado fibrosis pulmonar.
Debido a las similitudes y la alta homología entre el SARS-CoV-2 y el SARS-CoV, no es sorprendente que estos fenómenos, a saber, el aumento de la producción de citocinas y la fibrosis pulmonar, también sean evidentes en algunos casos graves de COVID-19. En conjunto, estas observaciones sugieren que la supresión viral de miR-16 puede contribuir al daño pulmonar en casos graves de COVID-19 como resultado de una mayor producción de ROS y TLR4. Por lo tanto, la regulación positiva de miR-16, quizás en forma de un imitador de miARN, puede ser una opción plausible para combatir los síntomas perjudiciales del COVID-19.
Resulta ver si estudios adicionales muestran que los sitios de unión predichos son realmente funcionales y si la esponja de miR-16 por SARS-CoV-2, SARS-CoV y MERS-CoV ocurre in vivo. Por último, es importante señalar que miR-16 está altamente conservado entre los mamíferos, lo que indica que miR-16 puede estar involucrado en un mecanismo de regulación del SARS-CoV-2 que existe en humanos y otros mamíferos, como los murciélagos, que puede ser infectado por CoV homólogos de murciélago.
Si estos HCoV de hecho han desarrollado un mecanismo que aproveche el potencial regulador del miR-16 en su anfitrión, el desarrollo de terapias que antagonicen esta interacción, como los inhibidores de miRNA para prevenir las interacciones del miR-16 con el genoma del SARS-CoV-2, es otra estrategia antiviral plausible.
Efectos potenciales de las interacciones del SARS-CoV-2 con el miR-200
Con las herramientas DIANA y TargetScan se predijo que miR-200b y miR-200c se unirían a una región altamente conservada de ACE2 3′-UTR, lo que sugiere que estos miRNA juegan un papel importante en la regulación de ACE2. Esto es particularmente importante en términos de infección por SARS-CoV-2, ya que tanto el SARS-CoV-2 como el SARSCoV utilizan el receptor ACE2 para la unión y la entrada en las células huésped.
La ACE2 también se considera un regulador negativo de la fibrosis pulmonar, ya que es responsable de la escisión de la angiotensina II (Ang II), una hormona peptídica que se ha relacionado con el desarrollo de fibrosis pulmonar mediante la activación de TGF-β. Estudios anteriores han demostrado que la regulación positiva de miR-200c-3p durante la infección por el virus de la influenza aviar A (H5N1) induce una disminución de los niveles de ACE2 y, en consecuencia, puede causar daño pulmonar. De manera similar a la infección por H5N1, se ha demostrado que el SARS-CoV-2 y el SARS-CoV inducen una disminución en la expresión de ACE2; sin embargo, no está claro si esto se debe a la unión de la proteína S al receptor o al resultado de la expresión de miR-200b / c regulada al alza. Como tal, se requiere más investigación in vitro e in vivo para determinar el perfil de expresión de miR-200b / c durante la infección por SARS-CoV y SARS-CoV-2. Además, la ECA2 se ha identificado como un importante regulador de la función cardíaca y se ha sugerido que la acumulación de Ang II puede provocar enfermedades cardíacas. Por lo tanto, investigaciones adicionales sobre la regulación de miR-200b / c de ACE2 puede proporcionar información útil sobre los factores de riesgo relacionados con el corazón y las complicaciones que ocurren en algunos pacientes infectados con SARS-CoV-2, como síndrome coronario agudo, miocarditis, arritmias y en casos graves, paro cardíaco.
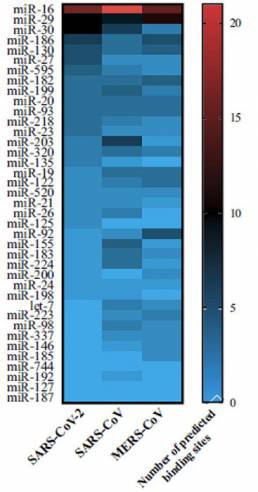
Figura 4 Número de sitios de unión de miARN predichos en los genomas de SARSCoV-2, SARS-CoV y MERS-CoV.
Funciones y vías de miR-24 que son susceptibles a la modulación por SARS-CoV-2
El sitio de unión de miR-24 predicho en la proteína S viral parece estar conservado entre los genomas de SARS-CoV-2, SARSCoV y MERS-CoV. Se ha demostrado previamente que miR-24 tiene un papel notable en la regulación de la furina, una serina proteasa que es importante para la activación proteolítica y la entrada en la célula huésped de algunos virus respiratorios, como el SARS-CoV-2, MERS-CoV, y H5N1. A menudo abundantes en varios tipos de tejidos, incluidos los pulmones, muchos virus respiratorios con envoltura utilizan la furina para escindir y modificar las glicoproteínas de superficie involucradas en la unión y fusión de la membrana, promoviendo así el potencial fusogénico del virus, que en última instancia puede provocar efectos sobre el tropismo celular y patogenicidad viral. Como existe un sitio de escisión similar a la furina (FCS) en la proteína SARS-CoV-2 S que no está presente en el SARS-CoV, hay motivos para creer que la furina puede contribuir a la patogenia e infección del SARS-CoV-2. De hecho, este FCS puede ser responsable de la alta actividad de fusión de membranas que es específica de SARS-CoV-2. Se ha demostrado que la furina activa el TGF-β1, que puede estar relacionado con el desarrollo de fibrosis pulmonar en casos graves de COVID-19. Por lo tanto, la posible regulación a la baja de miR-24 durante la infección por SARS-CoV-2 puede conducir tanto a un aumento de furina, lo que resulta en una mayor infectividad, como a un aumento de TGF-β1 que eventualmente puede conducir a daño pulmonar.
Por el contrario, un estudio encontró que el FCS y la activación por furina pueden no ser cruciales para la infección por SARS-CoV-2. Se determinó que el tratamiento de muestras infectadas con una alta concentración de proteasas similares a tripsina (HAT) de las vías respiratorias humanas restauró la capacidad fusogénica del SARS-CoV-2 sin FCS, lo que indica que el FCS y la presencia de furina pueden no ser completamente necesarios para Entrada del SARS-CoV-2 en las células huésped. En cambio, este estudio sugiere que la estructura general de la proteína S puede ser responsable de la alta actividad fusogénica del SARS-CoV-2, no la presencia del FCS; Sin embargo, sería necesaria más evidencia in vivo para apoyar esta hipótesis y descartar el posible papel.
Conclusión y direcciones futuras
En conclusión, los miARN representan objetivos nuevos y emergentes para la intervención terapéutica, incluso contra el SARS-CoV-2. Los objetivos celulares de los miARN se pueden suprimir agregando imitadores de miARN o se pueden regular al alza con el uso de anti-miR, de los cuales existen varias clases químicas. Los virus de ARN pueden interactuar y suprimir la función de los miARN endógenos, regular los miARN o incluso producir sus propios miARN en determinadas circunstancias. Se ha demostrado que contrarrestar estas interacciones evolucionadas es antiviral para otros virus como el VHC, donde los antagomir se han utilizado con éxito en ensayos clínicos. Dadas las similitudes y el tamaño del genoma del SARS-CoV-2, es razonable plantear la hipótesis de que tales estrategias también serán efectivas en el desarrollo de nuevas estrategias terapéuticas.
Tras un examen más detenido, se descubrió que varios miARN tenían sitios de unión potenciales en los genomas del SARS-CoV-2, SARSCoV y MERS-CoV. Entre ellos, se identificaron tres que se mostraron prometedores en la modulación de la infección por SARS-CoV-2, a saber, miR-16, miR-200 y miR-24. Muchos miARN humanos pueden ver una función alterada durante la infección aguda debido a un efecto de esponja del genoma del SARS-CoV-2 y la desrepresión resultante de los objetivos de ARNm. Es interesante considerar que algunas de estas interacciones pueden haber evolucionado como parte del tropismo viral como virus respiratorio y, por lo tanto, podría haberse producido una selección de cepas de virus para subvertir la señalización de miARN en las células huésped.
El trabajo actual y futuro se centra en obtener evidencia adicional para determinar si el SARS-CoV-2 realmente se basa en miARN específicos y a través de qué mecanismo. Un punto de partida sería validar los sitios de unión previstos de miARN específicos in vivo y determinar si estos sitios de unión son funcionalmente importantes para el ciclo de vida del SARS-CoV-2. Es probable que esto implique examinar características adicionales como la estructura secundaria del ARN, la abundancia de miARN y otras interacciones celulares. También resulta importante perfilar los cambios en la expresión de miARN durante la infección viral (es decir, qué miARN están regulados hacia arriba o hacia abajo), así como extender el análisis de detección de miARN para identificar miARN que se dirigen a las regiones UTR. La información sobre los efectos descendentes celulares que resultan de la expresión alterada de miARN también puede ser útil para comprender las complicaciones y síntomas asociados con la infección viral. También es importante investigar la conservación de los sitios de unión de miARN y los mecanismos reguladores entre otros mamíferos, ya que se han observado infecciones por CoV en otros mamíferos además de los humanos. Dado que algunos CoV pueden atravesar las barreras de las especies, comprender las interacciones y las regulaciones de miARN en estos genomas no humanos puede servir para mejorar nuestra comprensión de los posibles VHC emergentes como el SARS-COV-2.
En cuanto a la traducción de objetivos de miARN en terapias, es interesante observar que los miARN pueden administrarse mediante inyección o directamente en los pulmones utilizando tecnologías de aerosolización. Tanto los imitadores como los inhibidores se pueden administrar en cualquiera de estos formatos. Además, estas estrategias se pueden utilizar junto con la tecnología de ARN de interferencia pequeño (ARNip) para dirigirse directamente al genoma viral, lo que conduce a potentes terapias multicomponente basadas en ARN. Otras dependencias y susceptibilidades a la función de miARN también pueden surgir como estrategias antivirales contra el SARS-CoV-2. Por último, pueden utilizarse moduladores de moléculas pequeñas de la función de miARN para recapitular la función de miARN. Sin embargo, este último no puede ser eficaz sin que primero comprendamos las funciones de los miARN como dianas terapéuticas para la intervención y el tratamiento del SARS-CoV-2.
Referencia: Hum, C., Loiselle, J., Ahmed, N. et al. MicroRNA Mimics or Inhibitors as Antiviral Therapeutic Approaches Against COVID-19. Drugs (2021). https://doi.org/10.1007/s40265-021-01474-5
Extraído de:
https://pubmed.ncbi.nlm.nih.gov/33638807/
terapia microARN

Día de la obstetricia y la embarazada
¿Cómo se transforma la incertidumbre en una experiencia segura y asombrosa? Con una conexión única que hace de cada embarazo un viaje inolvidable.…
Los alimentos ricos en potasio mejoran la salud cardiovascular de las mujeres
De acuerdo con la Organización Mundial de la Salud (OMS), las enfermedades cardiovasculares (ECV) son la principal causa de muerte en todo el mundo y…
Viruela del mono: la OMS declara alerta máxima por el aumento de casos en el mundo
La Organización Mundial de la Salud ha declarado este sábado 23 de julio a la viruela de mono como una emergencia sanitaria internacional luego de un…
Los trastornos de ansiedad podrían transmitirse de padres a hijos y de madres a hijas
¿Difiere la probabilidad de transmisión de los trastornos de ansiedad de padres a hijos entre parejas de padres e hijos del mismo sexo y de sexo…




